この操作の前に、「Set Pathway Draw Data」を実行してください。IMC起動後一度だけ実行します。
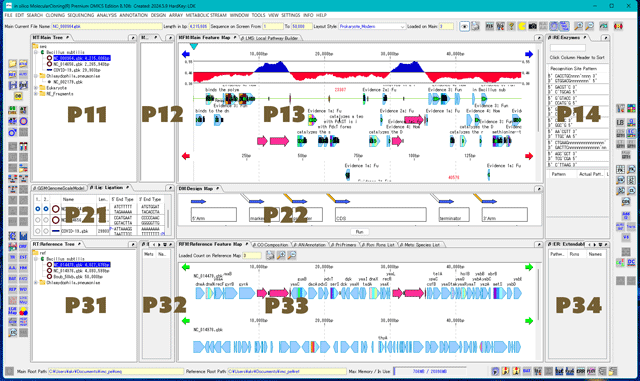
ローカルパスウェイビルダーで使用するペイン
P13: 上段3列目:LMS: Local Pathway Builder タブペイン
- 代謝パスウェイ描画領域です。ここに代謝パスウェイが描画されます。
- このタブペインは取り外し可能で、かつリサイズできます。
P21: 中段1列目:GSM: GenomeScaleModel タブペイン
P32: 下段2列目:EM: Extendable Metabolites タブペイン
- 現在描画されている代謝パスウェイから1反応で到達できるメタボライトのリストを表示する タブペインです。
P33:下段3列目:Rxn: Rxns List タブペイン
- Current GenomeScaleModel の Reaction List を表示する タブペイン です。
- 各カラムでソートできます。
P33: 下段3列目:Mets: Species List タブペイン
- Current GenomeScaleModel の Metabolte(Species) List を表示する タブペイン です。
P34: 下段4列目:ER: Extendable Rxns タブペイン
操作方法
- メニューから METABOLIC STREAM > Load GenomeScaleModel をクリックします。
- Load SBML ファイル選択ダイアログが表示されるので、ゲノムスケールモデル(SBMLフォーマット)を選択します。
- 注:現在使用できるゲノムスケールモデルは、BiGG モデルです。
- ロードが完了すると、P33: 下段3列目の Rxns List タブペインにその情報がリスト表示されます。
- 1反応の描画
- P33: Rxns List タブペインから最初に描画したい反応を選択し、クリックします。
- P13: Local Pathway Builder タブペインにその反応が描画されます。
- 現在描画されている反応の代謝物から続く反応を追加
- P32: Extendable Mets List タブペインに表示されている代謝物を1個クリックすると、その代謝物が仲介する反応をP34: Extendable Rxns List タブペインに表示します。
- P34: Extendable Rxns List の反応を1個クリックするとその反応がP13: Local Pathway Builderに追加されます。
