サイト内全文検索
サイト内ポピュラータグ
trouble
5
SNP
6
Qualifier
10
navigation
4
Mutation
5
Local Genome Rearrangement Map
6
Linear Map
9
License
5
Lane
9
Genome Design
7
Gene Cluster Alignment
8
Frame
6
Feature Map
6
Feature Layout Style
5
feature key
5
Feature
6
download
4
Codon
4
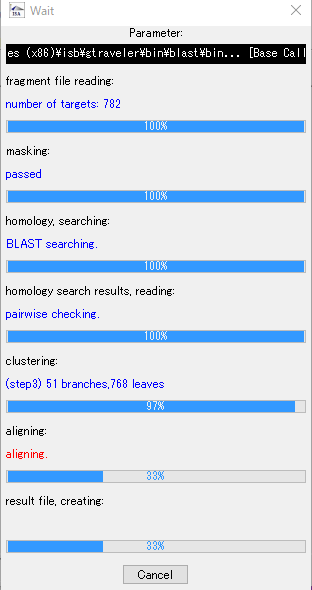
BLAST
11
Activation
4