- E04A1で説明したクエリー配列を直接入力する方法の他に、メインフィーチャーマップあるいはレファレンスフィーチャーマップ上に表示されているフィーチャー上でマウス右クリックすることにより、そのフィーチャ―の配列(塩基配列、あるいはCDS等であればアミノ酸配列も)をクエリー配列としてBlast検索を実行することができます。
- この検索では、ヒットしたサブジェクトとクエリー配列のフィーチャー付近のマップを自動的に表示し、相同領域同士をアラインメントするように表示することができます。
- クエリー配列とすることができるのは、メインフィーチャーマップ上のフィーチャーおよびメインフィーチャーマップ上でドラッグして選択された領域の塩基配列、あるいはその領域に存在するフィーチャーの塩基配列あるいはアミノ酸配列、およびレファレンスゲノムマップに表示されている任意のフィーチャーの塩基配列あるいはアミノ酸配列です。
- 検索対象となるデータベースは、メインフィーチャーマップ上のフィーチャーあるいは領域をクエリー配列とした場合は、レファレンスゲノムマップのゲノム塩基配列およびCDSフィーチャーなどの翻訳アミノ酸配列です。
- レファレンスゲノムマップ上のフィーチャーの塩基配列あるいはアミノ酸配列をクエリー配列とした場合は、カレントメインディレクトリ以下にあるゲノム塩基配列およびCDSフィーチャーなどに属するアミノ酸配列です。
- メインフィーチャーマップ上の1つのフィーチャーをクエリー配列として、カレントレファレンスディレクトリー上のゲノム塩基配列およびアミノ酸配列に対してBlast検索を実行します。
準備
- カレントメインディレクトリにいくつか近縁種ゲノム塩基配列ファイルをロードしておきます。
- カレントレファレンスディレクトリーにこれもいくつかの近縁種ゲノム塩基配列ファイルをロードしておきます。
- (IMCEEとIMCSEでは現在レファレンスゲノムマップに表示されている1つのゲノム塩基配列のみが対象となります。
操作
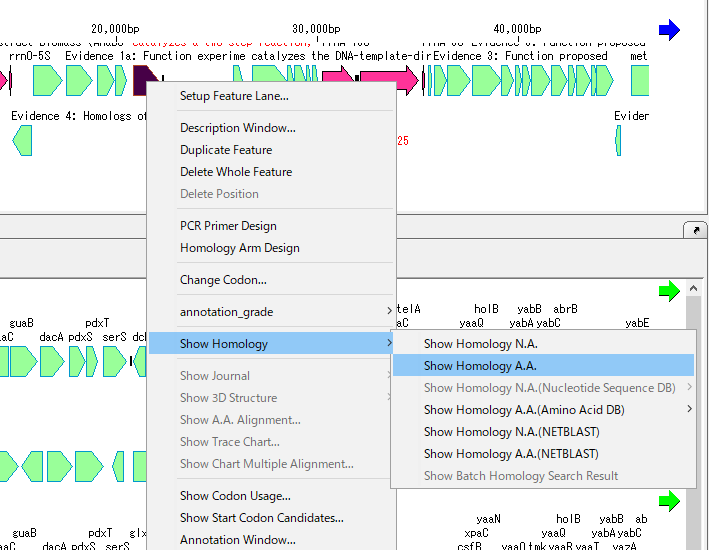
- メインフィーチャーマップ上の1つのCDSフィーチャー上でマウス右クリックします。
- メニューが表示されます。そのメニューからShow Homology -> Show Homology AAを選択します。
- 「Start Homology Search?」という確認メッセージが表示されます。
- 「はい(Y)」をクリックします。
- 相同性検索の実行が開始されます。実行中は進捗メッセージが表示されます。
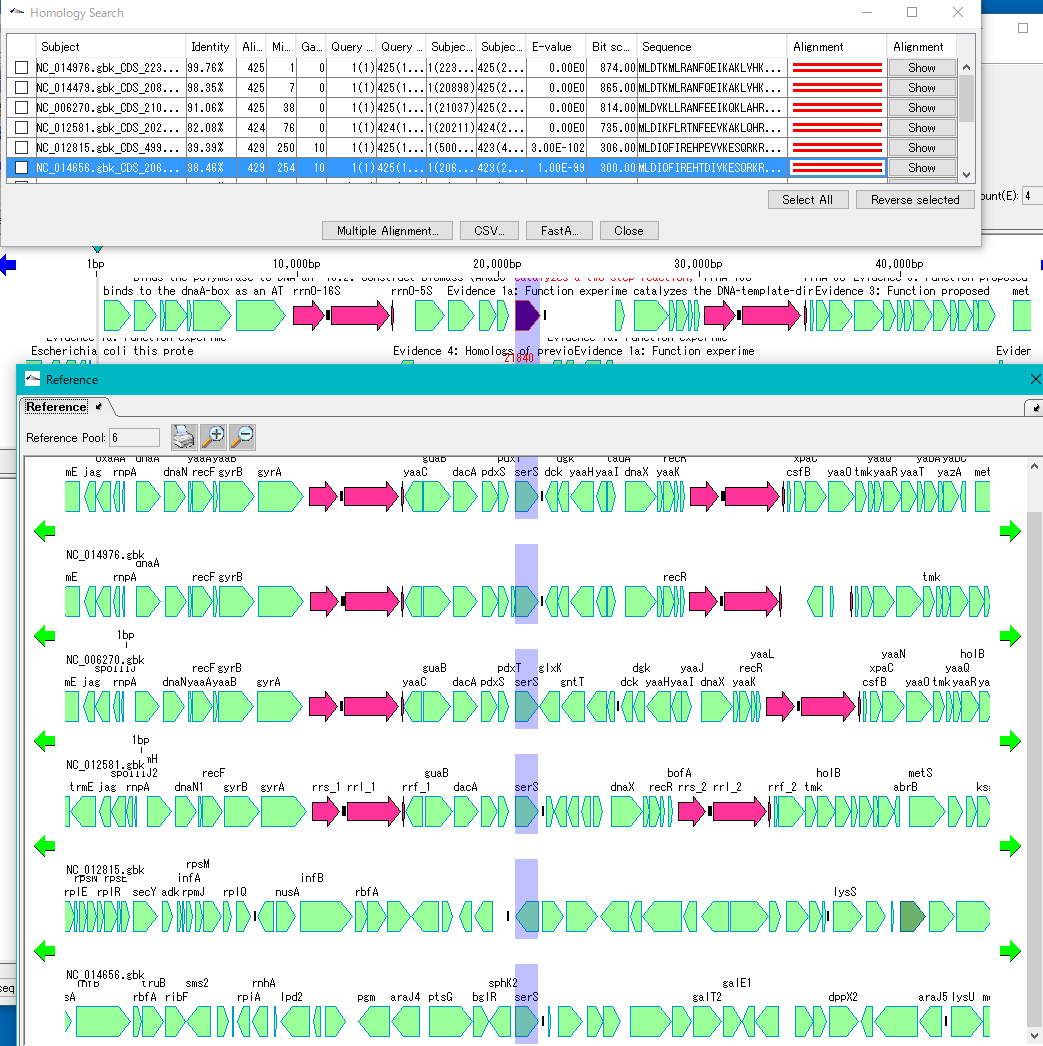
- 検索が完了すると「Homology Search」結果ダイアログが表示されます。
- このダイアログからいろいろな操作が可能です。
- 各データベースに対するヒットエントリの数はデフォールトでは1個になっているため、リストにはカレントレファレンスディレクトリーにロードされている配列数分のヒットしか表示されません。
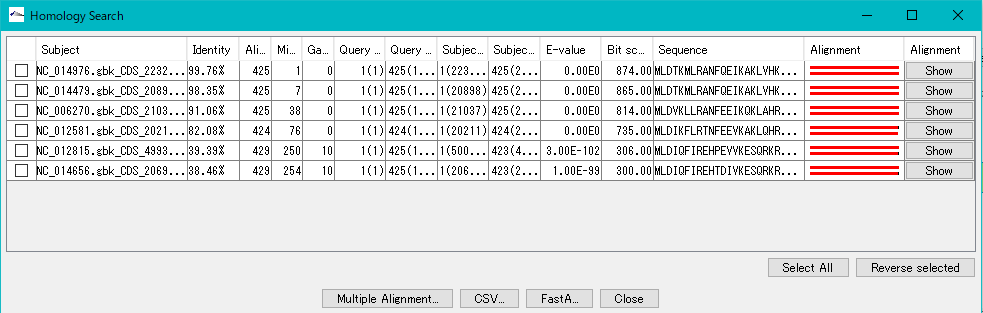
- トップヒットだけでなく、次点のヒットエントリも表示させたい場合は、検索を再実行し、「Show Parameter…」をクリックしてパラメータを表示し、以下のように「Maximum Number of Hits」、「-num_descriptions」および「-num_alignment」をそれぞれ2に設定します。
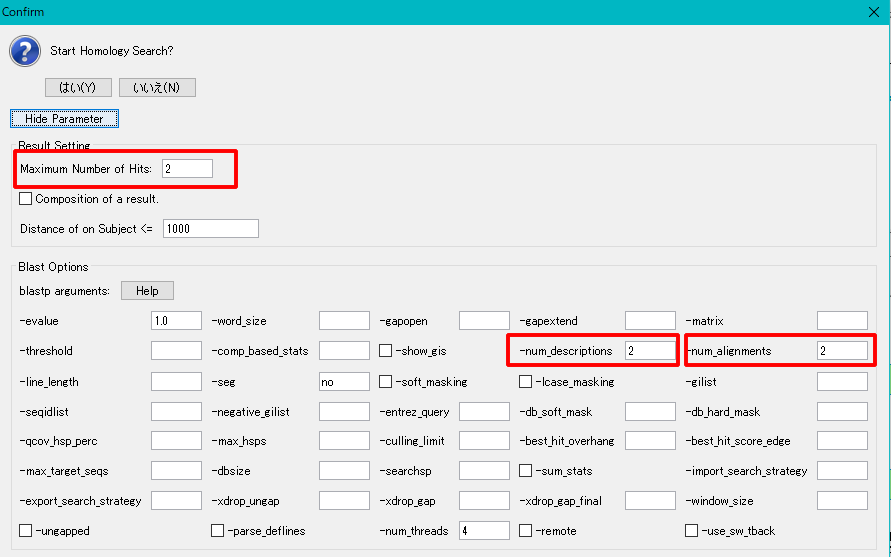
- 表示されるヒットの数は、必ずしも上記の数に一致するわけではありません。
- その他のクライテリアに従って、条件を充たすヒットが存在しない場合は、指定した数より少なく表示されます。
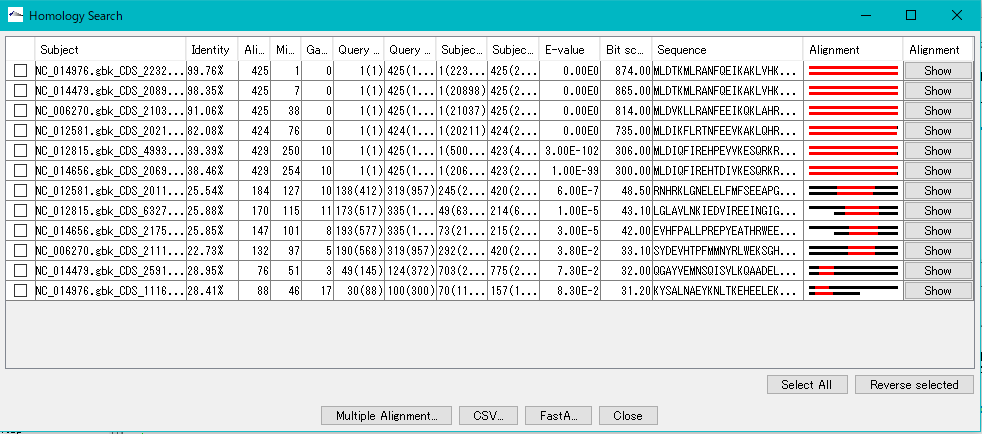
- ヒットエントリリストの1行をクリックします。ヒットエントリリストの1行をクリックします。
- すると、メインフィーチャーマップ上のクエリーフィーチャーとレファレンスマップのヒットエントリのフィーチャーがハイライトされ、それぞれのフィーチャーレーンがシフトして、並置されます。
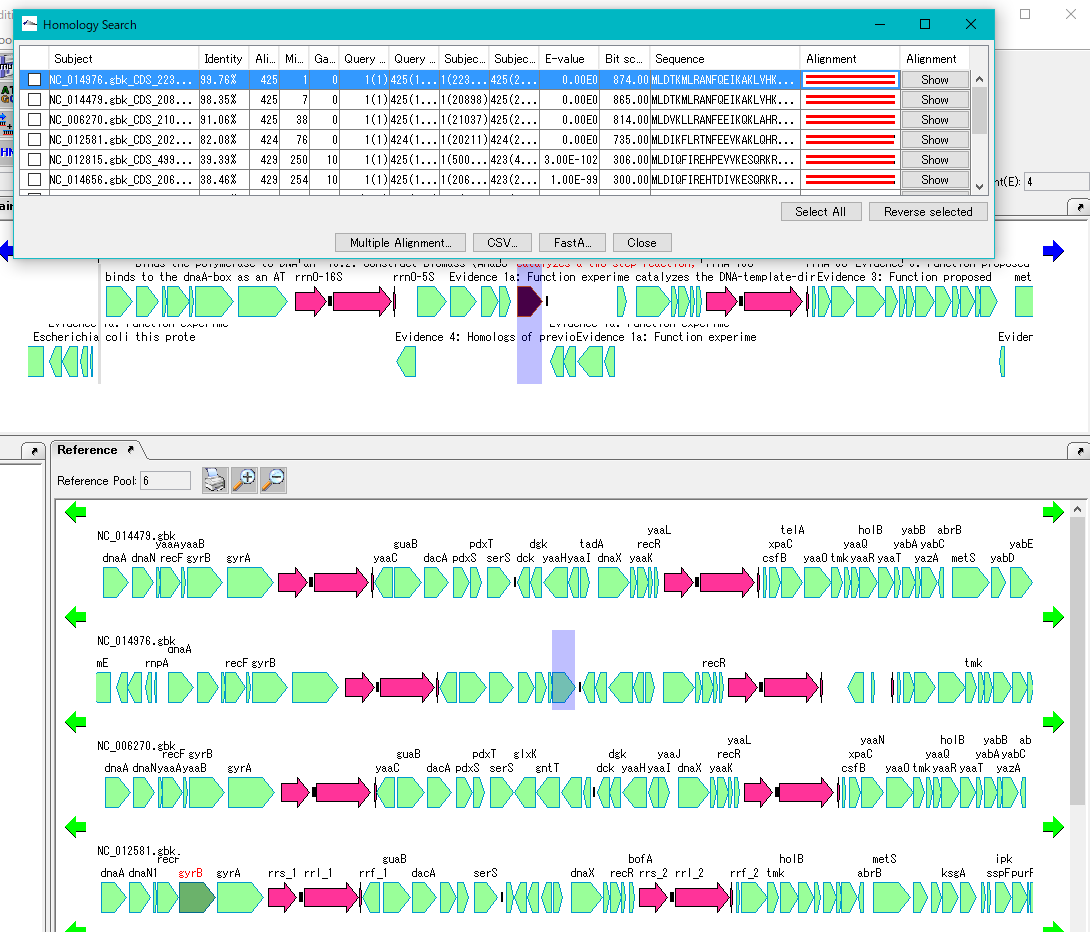
- 同様に他の行をクリックしていくと、レファレンスマップマップの各ゲノム配列上の相同フィーチャーが並置されます。
- ヒットエントリにチェックします。
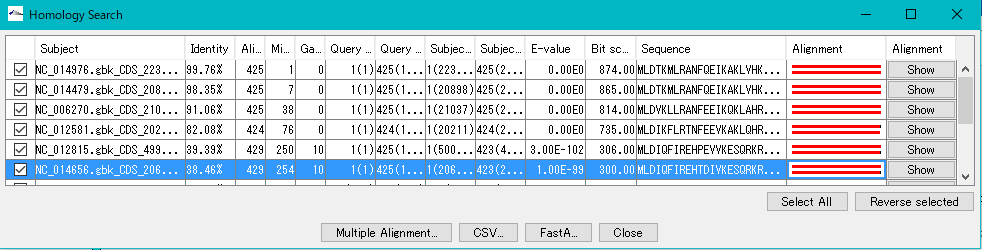
- 「Multiple Alignement…」をクリックします。「Multiple Alignement…」をクリックします。
- 「Start Multiple Alignment?」という確認メッセージが表示されます。
- 「Multiple Alignement」ダイアログが表示されます。
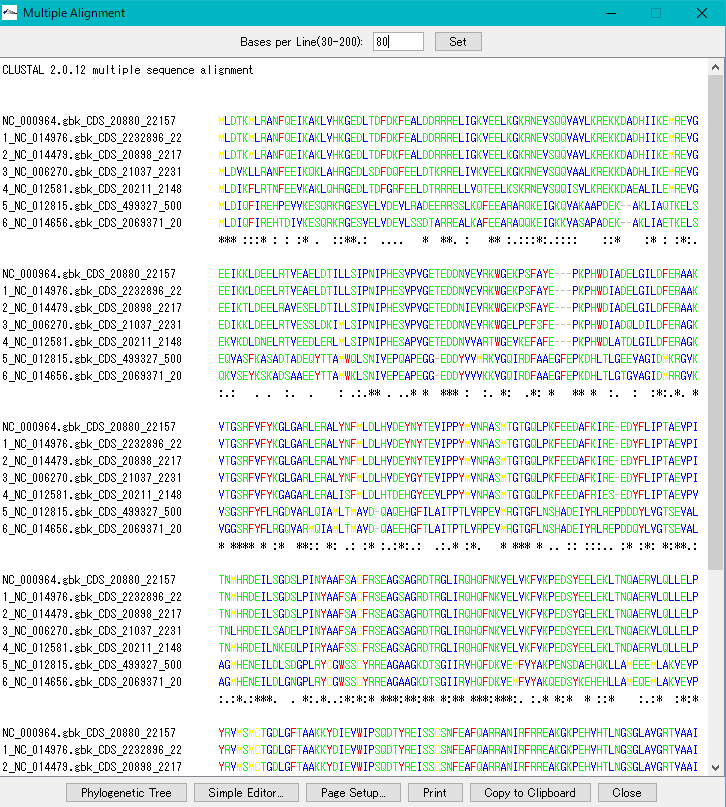
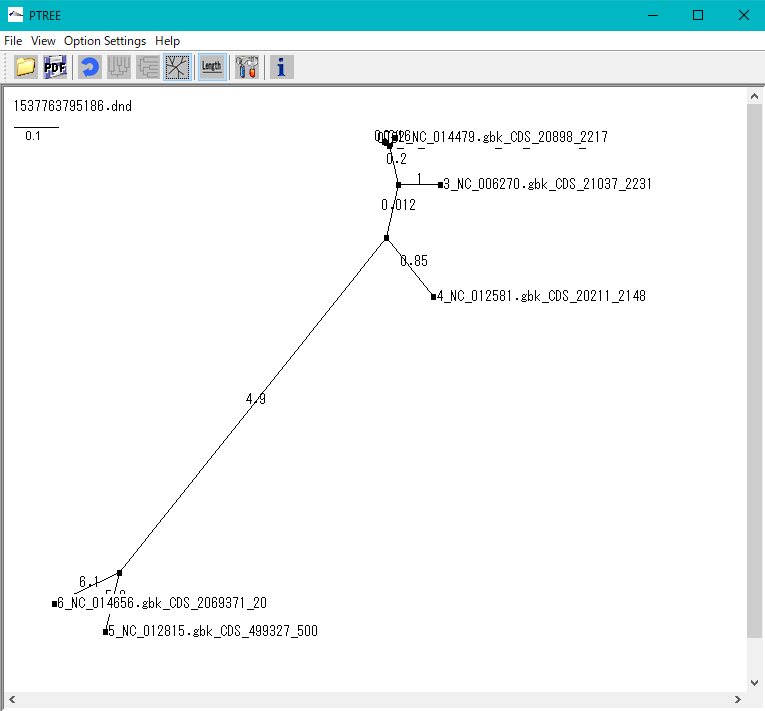
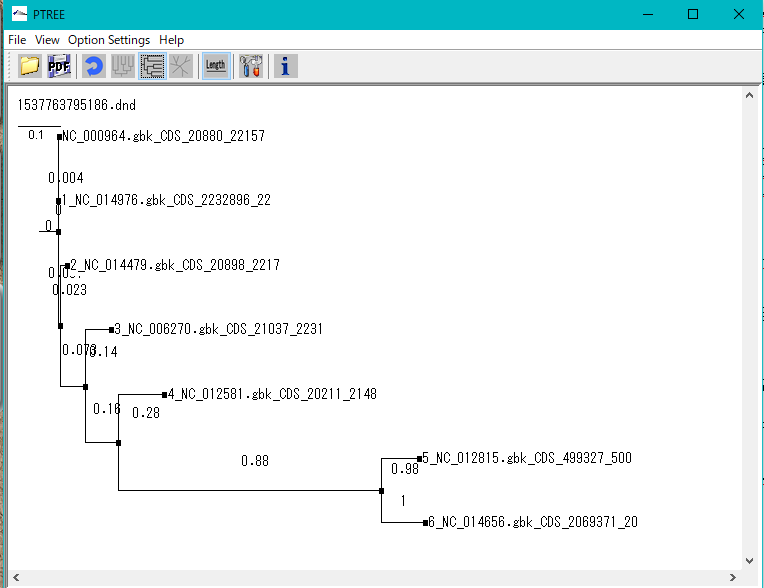
- この「Multiple Aligment」ダイアログからは分子系統樹も描画することができます。
- 「Phylogenetic Tree」をクリックします。
-